【摘要】:核酸DNA的分子量长度与其在凝胶中的迁移率有某种模型关系,经过实验发现其为非线性模型,现对某DNA标准品进行实验后得到如下数据,请试对其进行非线性回归分析。图10-1 DNA标准品电泳图分别拟合直线回归、多项式回归、对数回归、指数回归和幂函数回归,比较其回归模型参数。如果某未知DNA样品在与DNA标准marker相同条件进行电泳,其条带迁移率为0.4637,可根据模型预测计算分子量如下。
在生物学中核酸电泳是一个典型实验。核酸DNA的分子量长度(bp)与其在凝胶中的迁移率有某种模型关系,经过实验发现其为非线性模型,现对某DNA标准品进行实验后得到如下数据,请试对其进行非线性回归分析。

图10-1 DNA标准品电泳图


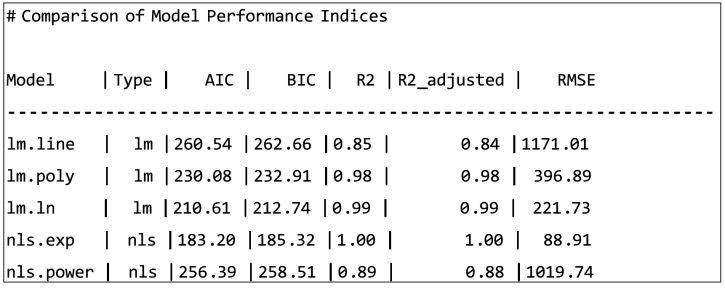
分别拟合直线回归、多项式回归、对数回归、指数回归和幂函数回归,比较其回归模型参数。


绘制四种非线性回归模型拟合曲线,判断实际拟合效果。

图10-2 二项式拟合
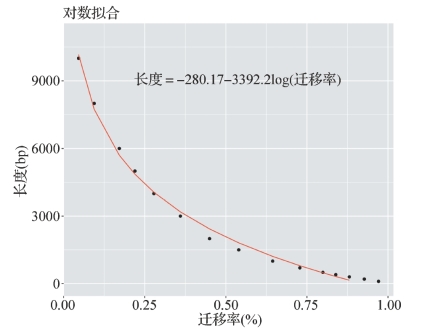
图10-3 对数拟合
 (https://www.xing528.com)
(https://www.xing528.com)
图10-4 指数拟合
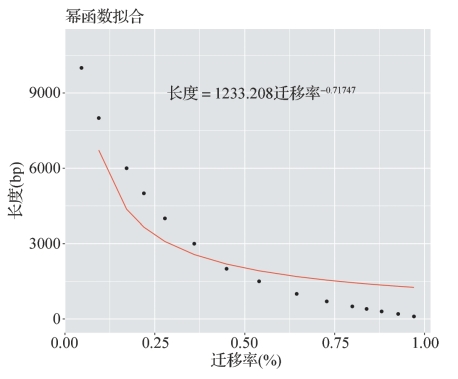
图10-5 幂函数拟合
比较对数回归和指数回归拟合模型的误差。


R包basic Trendline的通用函数trendline(x,y,model="")使用非常方便。可以一步完成绘图,添加线性或非线性拟合线,在图上显示回归方程及R2和回归模型的p值(不是参数的p值);并且它默认会同时输出模型summary()的结果。


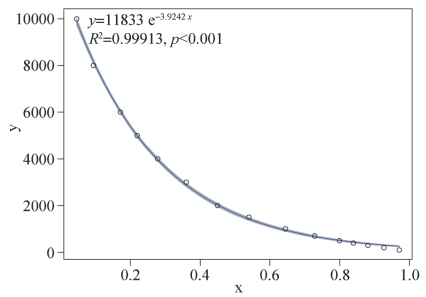
图10-6 使用basic Traidline包拟合
结论:在电泳实验中采用对数或者指数拟合模型最为正确。查阅文献,有研究者认为线性双链DNA分子在一定浓度琼脂糖凝胶中的迁移速率与DNA分子量的对数成反比,因此采用对数处理拟合更容易直观解释实验结果,但拟合精确度不如指数模型。如果某未知DNA样品在与DNA标准marker相同条件进行电泳,其条带迁移率为0.4637,可根据模型预测计算分子量如下。

免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。




